首页 > 心得体会 > 学习材料 / 正文
大豆蛋白含量主效位点qPRO-20-1的精细定位
2023-03-03 08:30:10 ℃杨 硕 武阳春 刘鑫磊 唐晓飞 薛永国 曹 旦 王 婉 刘亭萱 祁 航 栾晓燕,* 邱丽娟1,,*
大豆蛋白含量主效位点的精细定位
杨 硕1,3,**武阳春4,**刘鑫磊2唐晓飞2薛永国2曹 旦2王 婉3刘亭萱3祁 航3栾晓燕2,*邱丽娟1,3,*
1东北农业大学农学院, 黑龙江哈尔滨 150030;2黑龙江省农业科学院, 黑龙江哈尔滨 150030;3农作物基因资源与遗传改良国家重大科学工程 / 农业农村部种质资源利用重点实验室 / 中国农业科学院作物科学研究所, 北京 100081;4吉林农业大学生命科学学院, 吉林长春 130000
蛋白质含量是大豆重要的品质性状, 受多基因控制, 定位大豆蛋白质含量相关位点并挖掘候选基因, 对定向培育高蛋白含量大豆品种具有重要意义。本研究以优良品种黑农88作为母本与高蛋白优异种质P73-6B作为父本杂交, 构建了一个由265个单株组成的F2群体, 利用中豆芯1号对F2群体进行基因型鉴定并构建图谱, 结合蛋白质含量表型数据, 采用IciMapping 4.2软件在20号染色体上定位了一个QTL, 物理距离为2.46 Mb, 在区间附近筛选出11个多态性SSR标记并分析群体, 将定位区间从2.46 Mb缩小至100.8 kb。增加Gm20_28349696、Gm20_30805913、Gm20_31341532和Gm20_31483719共4个SNP位点, 进一步将区间缩小到95.8 kb。对区间内包含的4个基因的9个不同组织在Phytozome v13.1和PPRD RNA-seq 2个数据库中的表达量分析得到了2个候选基因, 分别为和基因, 本试验结果为大豆蛋白质含量基因克隆及蛋白质调控机制研究提供了理论基础, 为大豆高蛋白分子标记育种提供材料和技术支撑。
大豆; 蛋白质含量; QTL定位; 候选基因
大豆种子中富含蛋白质[1-2], 约占大豆籽粒的40%。大豆蛋白质拥有人类所需要的9种必需氨基酸, 是最为优良的植物蛋白之一, 可以满足人体的营养需求。大豆蛋白作为植物食用蛋白的主要来源, 在我国国民经济发展中有着重要的作用[3]。大豆蛋白含量是重要的经济性状, 我国大豆严重依赖进口, 对外依存度超过80%, 基本失去了制定大豆价格的主导权[4]。然而, 在我国经济高速发展的大背景下, 人们对大豆蛋白制品的需求却不断增加, 豆腐、豆腐干、豆浆、腐竹等高蛋白豆制品已经成为人们生活中的必需品[5], 为了改变现状掌握大豆市场的主动权, 需要提高大豆蛋白质含量, 改良大豆品种, 培育新的高蛋白优质品种。因此发掘调控大豆蛋白质含量的相关基因对于培育高蛋白大豆品种有着重要意义。
大豆蛋白质含量在大豆杂交后不能明确分组[6]且受环境因素影响很大, 因此对大豆蛋白质含量相关的基因挖掘难度较大。截止到2022年1月, 在SoyBase网站上(https://www.soybase.org/)有241个大豆蛋白质含量相关的QTL, 分布在20条染色体上。刘代铃等[7]利用‘南豆12’和‘九月黄’作为材料构建的RIL群体为材料在3个环境下共检测到10个蛋白相关QTL分布在9条染色体上, 表型贡献率为5.63%~9.68%。Huang等[8]利用华春2号和华耀作为亲本构建了大小为196个的F7:8-10重组自交系群体, 2种环境下在3号、6号、10号、13号、14号、18号共6条染色体上共检测到8个大豆蛋白含量相关QTL位点, 其中有5个QTL位点在2种环境下都能检测到, 可解释的表型变异为3.73%~17.69%。李曙光等[9]利用全基因组关联分析(GWAS)的方法检测到90个大豆蛋白相关QTL, 分布在20条染色体上, 共解释45.60%的遗传变异。张琦等[10]利用大豆染色体片段代换系将区间精细定位至852 kb, 区间内注释到15个基因, 并预测可能是蛋白质相关候选基因。虽然大豆蛋白质含量相关QTL已有众多报道, 但是没有克隆出相关基因, 精细定位研究也较少。
本研究通过200K芯片测序构建的图谱在20号染色体上定位获得大小为2.46 Mb的蛋白质含量相关QTL, 在此基础上利用F2群体的交换单株为材料将区间缩小至100.8 kb, 利用SNP和SSR分子标记数据和QTL IciMapping 4.2完备区间作图法(inclusive composite interval mapping, ICIM), 最终将区间缩小至95.8 kb, 为挖掘大豆蛋白质含量相关基因、解析相关基因的功能奠定理论基础。
1.1 群体构建和表型检测
本试验材料以综合性状良好的品种黑农88作为母本, 加拿大引进的高蛋白品种P73-6B作为父本构建F2分离群体共265株, 2019年在黑龙江省农业科学院大豆研究所种植, 并进行重组自交系(recombinant inbred lines, RIL)群体的构建。使用德国公司Bruker品牌傅里叶变光近红外光谱仪检测大豆蛋白质含量, 选择成熟且大豆表皮完好的籽粒进行蛋白质含量检测, 使用由实验室先前已构建完成的大豆蛋白含量检测模型, OPUS软件进行样品光谱数据分析, 每个样品检测3次重复, 取平均值为蛋白质含量。
1.2 基因组DNA提取
利用改良的CTAB法[11]提取基因组DNA, 取大豆新鲜叶片100 mg放入带有直径5 mm钢珠的1.5mL离心管中并浸入液氮中, 用高通量组织研磨仪研磨30 s, 加入600mL已加入巯基乙醇并摇匀的CTAB溶液, 在65℃水浴锅中水浴1.5 h, 取出后将其冷却至室温, 加入600mL酚氯仿并摇匀, 15,294´离心10 min。取上清液转入1.5mL离心管中加等体积异丙醇混匀, 放入-20℃冰箱20 min, 15,294´离心10 min, 弃上清液后加500mL 75%无水乙醇, 洗涤2次, 覆盖无菌纸晾干残留乙醇溶液, 加入300mL灭菌蒸馏水溶解DNA, 用1%琼脂糖凝胶电泳和紫外分光光度计检测DNA质量和浓度。
1.3 芯片测序和蛋白质含量QTL定位与验证
F2分离群体265个单株叶片样品送北京康普森生物技术有限公司进行200K芯片检测, 将基因型和蛋白质含量表型按照软件模板格式输入, 利用软件QTL IciMapping 4.2完备区间作图(inclusive composite interval mapping, ICIM)法[12], 对蛋白质相关的QTL进行定位, 将软件计算的1000次排列试验得到的95%分位数LOD值作为95%显著水平的LOD阈值, 在实际软件计算时得到的LOD值高于阈值时, 就认为此时的区间内存在一个QTL, 得到一个可解释贡献率为19.31%, 区间长度为2.46 Mb的QTL, 进一步缩短其区间长度, 得到区间长度为100.8 kb的QTL, 在此基础上通过7个标记结合F2群体大豆蛋白表型数据再次定位到该QTL, 并将其缩小至95.8 kb。
1.4 SSR标记筛选
在大豆数据库SoyBase (https://www.soybase. org/)网站上, 选择20号染色体上初定位区间内及附近的44对SSR标记, 根据引物序列信息, 北京擎科生物技术有限公司合成上述标记引物。利用母本黑农88和父本P73-6B进行引物多态性筛选, 得到11对亲本间差异SSR引物(表1)。使用的PCR反应体系为20mL, 体系包含2.0mL (20 ng µL–1) DNA、Easy酶2.0mL、10× Easybuffer 2.0mL、dNTPs 2.0mL、引物2.0mL (2.0mmol L–1)、无菌水10.0mL。PCR反应程序为95℃ 5 min; 95℃ 30 s, 55℃ 30 s, 72℃ 30 s, 34个循环; 72℃ 5 min, 4℃条件保存。用6%非变性聚丙烯酰胺凝胶电泳检测SSR标记的扩增产物。利用这11对标记对黑农88×P73-6B F2分离群体265个单株进行基因型鉴定, 其中与母本黑农88条带相同的记为A, 与父本P73-6B条带相同记为B, 对既有与父本相同也有与母本相同的条带记为杂合H, 条带缺失时记为-。
1.5 数据统计分析
利用Word Processing System计算群体蛋白质含量的最大值、最小值、平均数、标准差、峰度、偏度、变异系数和表达量差异图的绘制, SPSS19.0绘制蛋白质含量分布图并进行独立样本检验, R语言对定位区间两端标记SNP30805914、BARCSOYSSR_ 20_0649结合蛋白质含量表型数据进行差异性分析。
1.6 候选基因预测
在SoyBase (https://www.soybase.org/)网站上查询定位区间内基因的功能注释, 利用Phytozome网站在线查找并获得区间内基因的表达模式, 利用R语言软件生成基因表达热图, 利用PRRD网站查找并获得区间内基因的表达模式, 利用Word Processing System生成表达量图谱。
2.1 黑农88和P73-6B及其群体蛋白质表型分析
低蛋白母本黑农88蛋白质含量是43.67%, 高蛋白父本P73-6B蛋白质含量是49.64%, 双亲蛋白质含量呈现极显著差异(图1), 适合作为蛋白质含量QTL定位群体构建的亲本。由双亲杂交配制繁育获得的265个F2群体后代蛋白质含量分离较大, 最低为39.22%, 最高为53.25%, 平均值为47.01%, 变异范围在39.22%~53.25%之间, 变异系数为8.48% (表2)。265个F2蛋白质表型存在明显的超亲分离且呈现出连续的变异, 峰度为2.94>0 (峰度定义为四阶中心矩除以方差的平方减去3)、偏度0.26>0, 整体峰图呈现右偏尖峰分布, 与完全正态分布相比峰图更陡峭且右侧长尾, 说明在数据分布中, 含量中等和中等偏高蛋白质数据相对较多(图1), 为大豆蛋白质QTL的定位提供了基础。

表1 精细定位所用SSR标记

表2 亲本及F2群体蛋白质含量统计分析

图1 亲本及F2群体蛋白质含量频率分布直方图
垂直虚线表示两亲本表型的平均值和标准差。曲线代表密度图。***,<0.001。
Mean value and standard deviation of parental phenotypes are indicated by vertical dotted lines. Curve represents density plot. ***,< 0.001.
2.2 基于200K芯片的蛋白质含量相关QTL定位
将F2群体265个单株的基因型结合蛋白质含量表型数据, 利用软件QTL IciMapping 4.2完备区间作图法对蛋白质含量相关的QTL进行定位, 对20号染色体进行遗传图谱构建, 共有1350个SNP标记输入软件, 筛选删除不连锁标记和冗余标记, 最终使用266个SNP标记作图, 图距152.97 cM, 标记间平均距离0.57 cM, 在20号染色体上得到1个QTL位点(图2), 位于Gm20_28349696和Gm20_30805913之间, LOD值为12.23, 阈值LOD值为2.5, 负的加性效应说明高蛋白等位基因来自父本P73-6B, QTL定位区间大小约2.46 Mb, 可解释表型遗传变异19.31% (表3), 包含42个基因。
2.3 蛋白质含量QTL精细定位
在定位区间内及附近选择SSR标记, 共选择BARCSOYSSR_20_0606至BARCSOYSSR_20_0649之间的44个SSR标记, 筛选出19个亲本间具有多态性SSR标记, 选择其中11个条带清晰的SSR标记分析群体(表1)。将QTL区间定位至BARCSOYSSR_ 20_0647至BARCSOYSSR_20_0649之间(图3), 进一步缩短其区间长度, 得到区间长度为100.8 kb的QTL, 命名为, 区间内含有5个基因。为了进一步缩小定位区间, 利用200K芯片测序数据, 筛选在定位区间内及附近的亲本间存在多态性差异的SNP标记位点并获取该位点F2群体基因型, 共筛选得到4个SNP标记位点, 分别为Gm20_28349696、Gm20_30805913、Gm20_31341532和Gm20_31483719。挑选区间内3个已完成F2群体基因型分析鉴定的SSR标记BARCSOYSSR_20_0636、BARCSOYSSR_20_0647, 结合这7个标记位点的F2群体基因型和大豆蛋白质含量数据, 对QTL进行精细定位, 在100.8 kb大小的区间基础上(图3), 进一步将定位区间缩小至Gm20_30805913–BARCSOYSSR_20_0649之间(图4),大小为95.8 kb (图4), 区间内包含4个基因, 表型贡率由19.31%提升至21.44% (表4)。

图2 用黑农88和P73-6B构成的F2群体在大豆20号染色体定位蛋白质QTL

表3 在黑农88×P73-6B F2群体鉴定到的QTL

图3 蛋白含量QTL qPRO-20-1的精细定位
A: 利用HN88×P73-6B F2200K芯片测序数据遗传图谱构建定位的QTL位点在SNP2839696~SNP30805913之间。B: 利用BARCSOYSSR_20_0608、BARCSOYSSR_20_0613、BARCSOYSSR_20_0616、BARCSOYSSR_20_0618、BARCSOYSSR_20_0619、BARCSOYSSR_20_0629、BARCSOYSSR_20_0636、BARCSOYSSR_20_0638、BARCSOYSSR_20_0644、BARCSOYSSR_20_0647、BARCSOYSSR_20_0649共11个标记鉴定5个交换单株(297、272、68、72和209)的基因型, 并验证并初定位。C: 利用芯片数据SNP位点标记28349696、30805913、31343532、31483719和3个SSR标记BARCSOYSSR_20_0636、BARCSOYSSR_20_0647、BARCSOYSSR_20_0649将区间精细定位至标记30805913~BARCSOYSSR_20_0649之间。D: 获得4个候选基因、、、。
A: construct a genetic map to locate the QTL sitebetween SNP 2839696–SNP 30805913 using Heinong 88×P73-6B F2200K chip sequencing data. B: the genotypes of five exchange monocultures (297, 272, 68, 72, and 209) were identified using a total of 11 markers BARCSOYSSR_20_0608, BARCSOYSSR_20_0613, BARCSOYSSR_20_0616, BARCSOYSSR_20_0618, BARCSOYSSR_20_0619, BARCSOYSSR_20_0629, BARCSOYSSR_20_0636, BARCSOYSSR_20_0638, BARCSOYSSR_20_0644, BARCSOYSSR_20_0647, BARCSOYSSR_20_0649; the genotypes were verified and primed. C: the interval was finely localized to the SNP marker 30805913–BARCSOYSSR_20_0636, BARCSOYSSR_20_0649 using the chip data bits SNP locus markers 28349696, 30805913, 31343532, 31483719, and the three SSR markers (BARCSOYSSR_20_0636, BARCSOYSSR_20_0647, BARCSOYSSR_20_0649). D: four candidate genes of,,, andwere obtained.
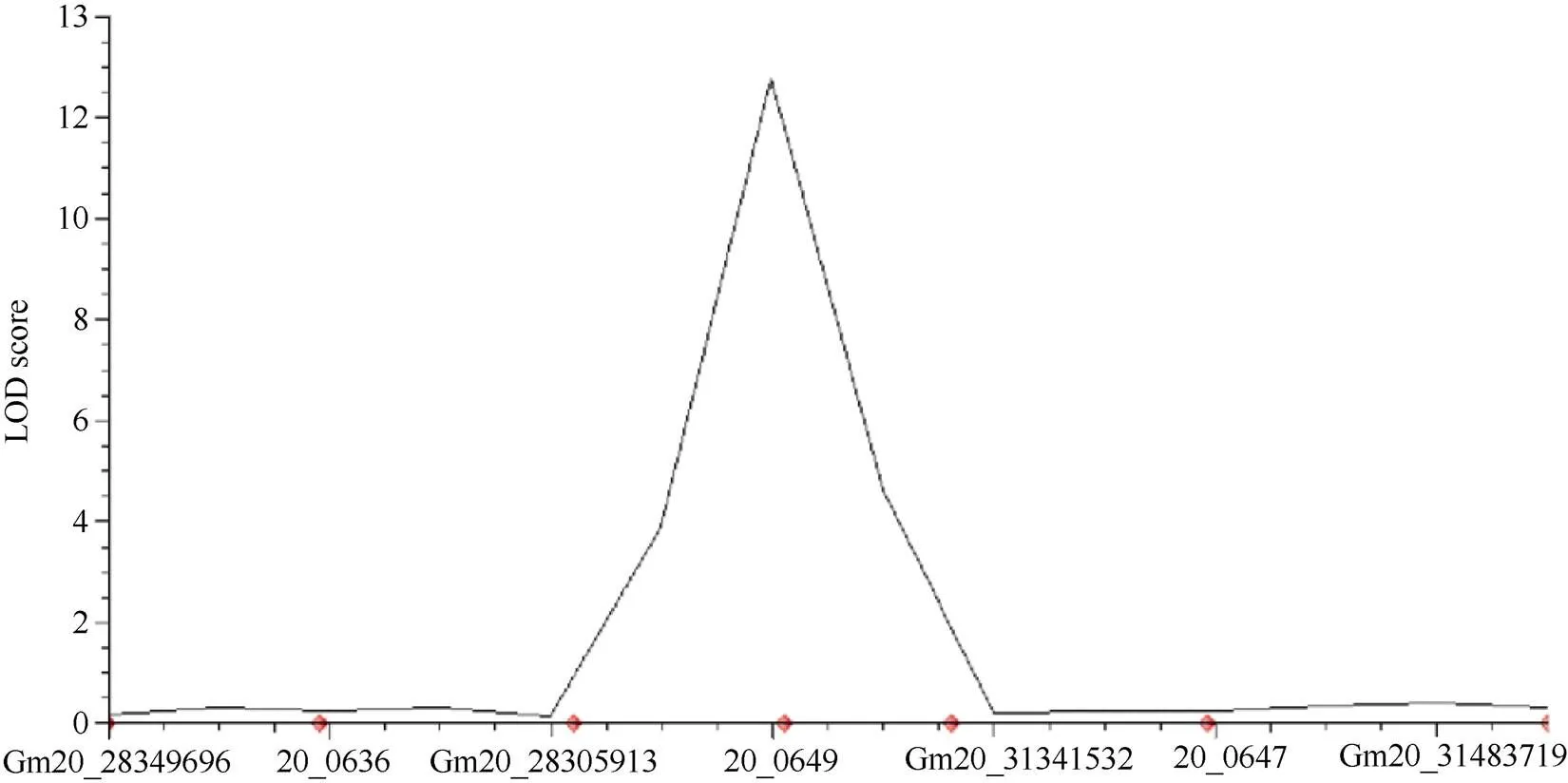
图4 利用7个分子标记定位黑农88×P73-6B大豆F2群体20号染色体的蛋白质QTL

表4 在黑农88×P73-6B F2群体验证得到的QTL
2.4 qPRO-20-1位点两端标记蛋白质表型和基因型差异显著性分析
对F2群体位点两侧的2个基因型标记与蛋白质表型(图5)进行分析, Gm20_30805913标记位点具有黑农88 (A)基因型的有62个, 蛋白质表型含量范围为40.01%~52.36%, 平均值为44.38%。Gm20_ 30805913标记具有P73-6B (B)基因型的有67个, 蛋白质表型含量范围为43.90%~52.90%之间, 平均值为47.95%。BARCSOYSSR_20_0649标记具有黑农88 (A)基因型的有60个, 蛋白质表型含量范围在40.02%~ 52.35%, 平均值为44.33%。BARCSOYSSR_20_0649标记位点具有P73-6B (B)基因型的有68个, 蛋白质表型含量范围在43.67%~52.90%之间, 平均值为47.89%。2个标记位点不同等位基因的蛋白质含量呈现极显著差异, 这也说明这个标记与蛋白质含量紧密相关。
2.5 候选基因预测分析
根据区间定位结果在SoyBase (https://www. soybase.org/)网站上搜查, 在区间内有4个基因(表5), 在Phytozome v13.1网站查找4个基因在9个不同时期及部位的表达模式, 利用R语言软件生成对基因在不同组织中的表达量热图(图6)。和基因在大豆茎、花、根毛、叶片、根瘤、荚、种子、顶端分生组织以及根中均有表达。基因在大豆9个不同的时期及部位均无表达。基因只在大豆种子中有表达。大豆蛋白质是大豆籽粒中重要的组成成分, 蛋白质来源大豆籽粒,、和基因均在大豆种子中有表达, 但表达以基因最高为16.86, 以基因最低为1.60, 表达量大小为>>, 在PPRD RNA-seq数据库中(http://ipf.sustech.edu.cn/pub/plantrna/), 查找区间内4个基因在9个不同组织的表达量(图7), 数据分析发现, 在和基因中9个时期的表达量都较高,基因在这9个组织的表达量都极低,基因在9个不同的时期及部位几乎无表达。在大豆籽粒中基因表达量19.21>基因15.55, 结合2个数据库的结果分析, 初步确定基因和基因可能与大豆蛋白质含量有关。

图5 F2群体中qPRO-20-1位点两侧标记Gm20_30805913与BARCSOYSSR_20_0649的基因型与蛋白质表型相关性分析
A: Gm20_30805913的基因型与表型相关性分析; B: BARCSOYSSR_20_0649的基因型与表型相关性分析。图中横坐标为基因型, 低蛋白为a, 高蛋白为b, 纵坐标为蛋白质含量。***:< 0.001。
A: genotype and phenotype correlation of Gm20_30805913; B: genotype and phenotype correlation of BARCSOYSSR_20_0649. The abscissa is the genotype, the high protein is denoted as b, the low protein is denoted as a, and the vertical ordinate is protein content. ***:< 0.001.

表5 定位区间内候选基因注释

图6 用Phytozome v13.1获得4个基因的表达谱

图7 用PPRD数据库获得4个基因在大豆9个不同组织的表达量
3.1 大豆20号染色体蛋白质含量QTL定位
大豆蛋白质含量相关QTL报道较多, 到2021年1月7号为止, 在SoyBase (https://www.soybase. org/)网站上已公布了241个QTL, 分布在20条染色体, 其中20号染色体定位蛋白质含量位点的数量最多, 有24个, 9个QTL的定位区间包含本研究得到的位点。分别是Seed protein 1-1 (25,275,083~34,302,228) 9.03 Mb、Seed protein 1-2 (24,177,772~34,302,228) 10.12 Mb、Seed protein 15-1 (27,664,504~33,590,931) 5.93 Mb、Seed protein 30-1(2,047,603~35,362,576) 33.31 Mb、Seed protein 31-1 (8,169,252~33,590,931) 25.42 Mb、Seed protein 34-11 (25,498,552~35,362,576) 9.86 Mb、Seed protein 36-26 (2,615,587~41,195,418) 38.58 Mb、Seed protein 37-8 (3,725,849~34,052,339) 30.33 Mb、Seed protein 39-4 (25,275,083~34,052,339) 9.78 Mb。在这9个QTL定位区间内最大的为Mao等[13]定位的38.58 Mb, 最小的为Chung等[14]定位的5.93 Mb。魏荷等[15]通过查询整理蛋白质含量相关QTL并对其进行连锁遗传绘图发现, 在20号染色体278,844,457~31,972,955 bp区段的蛋白质QTL位点检测频率最高, 本试验定位得到的QTL在大豆蛋白质QTL定位数量最多的20号染色体, 所在的区段又是在定位热点区段, 也间接证明了本试验定位结果的正确性以及具有的重要研究价值。
3.2 大豆蛋白质含量QTL精细定位
大豆蛋白质含量是典型的数量性状, 受到多基因控制[16], 尽管前人对大豆蛋白质含量相关的QTL进行大量研究, 但关于蛋白质含量相关QTL的精细定位研究较少。目前蛋白质含量QTL精细定位主要有2种方法, 一种是利用构建传统遗传群体的方式, 常见重组自交系(RIL)群体、染色体片段置换系(chromosome segment substitution lines, CSSL)群体等永久性群体的方式进行QTL精细定位, 如武阳春等[17]通过构建RIL群体结合BSA构建混池的方式在19号染色体定位到大小为384 kb的新位点, 并有2个候选基因; Yang等[18]通过染色体片段置换群体, 将QTL精细定位在329 kb区间内, 区间内包含42个基因。另一种是利用自然群体全基因组关联分析的方式, 如Zhang等[19]利用GWAS分析在15号染色体基因间区得到一个38.7 kb的区间。而本研究利用200K芯片获得基因型数据进行图谱构建, 在20号染色体上得到的2.46 Mb的初定位区间并结合F2群体和SSR分子标记将区间缩小至95.8 kb, 可解释21.44%的遗传变异, 该QTL是目前在20号染色体定位区间最小贡献率较大的主效QTL, 位点基因也与已报到的不同, 研究结果为大豆蛋白质相关主效基因的挖掘和克隆奠定了基础。
3.3 大豆蛋白质含量候选基因
大豆蛋白质的合成代谢是一个复杂的过程, 有2条主要的代谢途径, 一条是氮素和碳素的合成, 是调控氨基酸合成的代谢途径[20], 在该途径中, 大豆吸收空气中的氮气并通过谷氨酰胺合成酶等调控模式将其转化为氨基, 并为氨基酸合成提供原料[21], 而氨基酸又是蛋白质合成的基本单位。氨基酸的种类多样, 多数都有自己的合成途径, 在合成通路中受到众多酶和基因的影响, 具体的反应发起位点, 在组织细胞中如何发生变化, 目前尚无定论[22]。另一条是储藏蛋白基因的合成转录、翻译和加工[23]。大豆储藏蛋白是大豆种子总蛋白中的主要组成部分,主要分为4种包括2S、7S、11S、15S蛋白[24], 其中大豆种子7S和11S又是种子储藏蛋白的主要成分, 约为70%。它们的合成方式略有不同, 主要途径是经过颗粒型内质网合成前体、高尔基体分选、囊泡运输、液泡中贮存通过酶加工等方式转化合成为成熟贮藏蛋白质[25-28]。
因为大豆蛋白质含量与大豆种子时期的相关基因表达量有关联, 所以对本研究得到的4个基因不同组织进行表达量分析, 利用Phytozome v13.1网站和R语言软件获得表达量并生成热图, 结果表明、和这3个基因在大豆的种子发育时期表达, 可能与蛋白质含量有关。
基因注释表明(表4),是PantherFam黄嘌呤尿嘧啶/维生素C渗透酶家族成员之一, 又属于NCS2或NAT家族, 已预测有2000个成员以上, 在主要的生物类群中均有分布[29], 其中仅有较少成员在功能上被鉴定, 如在大肠杆菌中的黄嘌呤通透酶XanQ经常被用作为极性残基的诱变底物, 尿嘧啶同源物的研究[30], 尿酸/黄嘌呤通透UAPA的缺乏转运不影响其对黄嘌呤的亲和力但可降低尿酸并可导致与其他嘌呤相错误结合几率增加[31]。而在拟南芥的同源物中进行的鉴定, 由于效能低等原因, 功能暂时未知[32]。尚无功能预测相关报道。是带有RNA结构域的NTF2超家族蛋白, NTF2超家族蛋白是一组有共同折叠的通用蛋白结构域, 没有共同的基序, 这些结构域在不同蛋白质中具有不同的功能[33]。据报道[34]在拟南芥中NTF2蛋白参与物质从细胞质到细胞核的运输过程, 通过识别核定位信号(NLS)来输入核蛋白, 也在纺锤体形成、核膜组件等起到作用[35-36], 在小麦中通过对NTF2沉默降低了小麦条锈病的抗性[37]。其同源基因在蓝藻的氮素和光合作用相关途径中被预测为microRNAs (miRNA)的靶基因之一[38], 而大豆蛋白质的基本组成单位氨基酸, 其氨基的来源又是与氮素有关, 该基因是否与是大豆蛋白含量相关候选基因还需进一步验证。
3.4 大豆高蛋白种质筛选
大豆蛋白质含丰富的营养, 价值颇高, 高蛋白大豆种质筛选在育种中具有重要的意义[39]。我国大豆主要是食用, 高蛋白育种是重要的方向。本研究所用母本黑农88和父本P73-6B蛋白质含量均大于国家(省)品种审定所划定的高蛋白(43%)的界限, 且差异大于6%, 母本F2群体蛋白质含量均值高达47.01%, 变异范围广达14.03%, 超亲分离现象明显, 高于43%的优异高蛋白含量个体有111个, 约占42%。这个群体不仅仅是一个定位群体, 更是一个难得的高蛋白育种群体。相比于其他构建蛋白质群体定位的研究, 如闫海波等[40]所构建的群体双亲蛋白质含量分别为40.20%和42.20%, 变异范围为2.2%, 后代群体蛋白质含量平均值为40.7%, 变异范围9.9%。Huang等[41]等利用蛋白质含量41.5%的Huachun 2和42.9% Wayao构建了分离群体, 其后代群体在4个环境下蛋白质含量平均值最低为42.9%, 最高为45.6%, 平均为43.7%, 平均变异范围为7.8%。Kaleri等[42]利用Heihe 36 (蛋白质含量39.80%)和Dongnong L13 (蛋白质含量45.50%)、Henong 60 (蛋白质含量38.47%)和Dongnong L13 (蛋白质含量45.50%), 分别组成2个重组自交系, 后代群体的平均蛋白质含量为42.78%, 蛋白质含量最小值为37.67%, 最大值为45.47%, 变异范围为7.80%。本试验所构建的群体相比上述这3个群体, 双亲蛋白质含量更高且亲本间蛋白质含量差距也较大, 在后代群体中蛋白质含量的平均值更高、变异范围更广, 在总体的蛋白质含量和分布上在高蛋白育种上更有优势。该群体不仅包含本研究定位的QTL外, 还包括其他QTL, 利用分子标记聚合多个高蛋白QTL, 有助于培育高产高蛋白新种质和新品种。
利用黑农88×P73-6B构建的265个F2分离群体进行200K芯片分析, 获得基因型数据并构建遗传图谱, 在20号染色体定位到一个大小为2.46 Mb的大豆蛋白质相关的QTL, 通过定位区间引物加密, 进一步缩短区间长度, 得到区间长度为100.8 kb的QTL, 在此基础上通过7个标记结合F2群体大豆蛋白表型数据再次定位到该QTL, 最终将QTL缩小至Gm20_30805913–BARCSOYSSR_20_0649之间, 大小为95.8 kb, 贡献率为21.44%。对区间内包含的4个基因的9个不同组织, 在Phytozome v13.1和PPRD RNA-seq两个数据库中的表达量分析, 得到了2个候选基因, 分别为、。
[1] Leamy L J, Zhang H Y, Li C B, Chen C Y, Song B H. A genome-wide association study of seed composition traits in wild soybean ()., 2017, 18: 18.
[2] 陈静静, 刘谢香, 于莉莉, 卢一鹏, 张嗣天, 张昊辰, 关荣霞, 邱丽娟. 利用BSA法发掘野生大豆种子硬实性相关QTL. 中国农业科学, 2019, 52: 2208–2219.
Chen J J, Liu X X, Yu L L, Lu Y P, Zhang S T, Zhang H C, Guan R X, Qiu L J. Mining QTLs related to seed firmness of wild soybean by BSA method., 2019, 52: 2208–2219 (in Chinese with English abstract).
[3] 邱丽娟. 大豆高蛋白育种的研究概况与展望. 作物杂志, 1990, (2): 3–5.
Qiu L J. Research situation and prospect of soybean high protein breeding., 1990, (2): 3–5 (in Chinese with English abstract).
[4] 时玉强, 鲁绪强, 马军, 刘军, 刘汝萃. 大豆蛋白在传统豆制品中的应用. 中国油脂, 2017, 42(3): 155–157.
Shi Y Q, Lu X Q, Ma J, Liu J, Liu R C. Application of soybean protein in traditional soybean products., 2017, 42(3): 155–157 (in Chinese with English abstract).
[5] Liu Z H, Zhao J H. Research progress of soybean protein., 2019, 4: 69–72.
[6] Teng W, Lei F, Wen L, Wu D, Xue Z, Han W, Li W. Dissection of the genetic architecture for soybean seed weight across multiple environments., 2017, 68: 358–365.
[7] 刘代铃, 谢俊锋, 何乾瑞, 陈四维, 胡跃, 周佳, 佘跃辉, 刘卫国, 杨文钰, 武晓玲. 净作和套作下大豆贮藏蛋白11S、7S组分相对含量的QTL分析. 作物学报, 2020, 46: 341–353.
Liu D L, Xie J F, He Q R, Chen S W, Hu Y, Zhou J, She Y H, Liu W G, Yang W Y, Wu X L. QTL analysis of relative contents of 11S and 7S components of soybean storage protein under net cropping and intercropping., 2020, 46: 341–353 (in Chinese with English abstract).
[8] Huang J H, Ma Q B, Cai Z D, Xia Q J, Li S X, Jia J, Chu L, Lian T X, Nian H, Cheng Y B. Identification and mapping of stable QTLs for seed oil and protein content in soybean [(L.) Merr.]., 2020, 68: 6448–6460.
[9] 李曙光, 曹永策, 贺建波, 王吴彬, 邢光南, 杨加银, 赵团结, 盖钧镒. 大豆巢式关联作图群体蛋白质含量的遗传解析. 中国农业科学, 2020, 53: 1743–1755.
Li S G, Cao Y C, He J B, Wang S B, Xing G N, Yang J Y, Zhao T J, Gai J Y. Genetic analysis of protein content in soybean population based on nested association mapping., 2020, 53: 1743–1755 (in Chinese with English abstract).
[10] 张琦, 尹彦斌, 蒋洪蔚, 张维耀, 潘校成, 武小霞. 大豆子粒蛋白质含量QTL的精细定位. 分子植物育种, 2019, 17: 8152–8157.
Zhang Q, Yin Y B, Jiang H W, Zhang W Y, Pan X C, Wu X X. Fine mapping of QTL for protein content in soybean kernel., 2019, 17: 8152–8157 (in Chinese with English abstract).
[11] Porebski S, Bailey L G, Baum B R. Modification of a CTAB DNA extraction protocol for plants containing high polysaccharide and polyphenol components., 1997, 15: 8–15.
[12] Meng L, Li H H, Zhang L Y, Wang J K. QTL IciMapping: integrated software for genetic linkage map construction and quantitative trait locus mapping in biparental populations., 2015, 3: 269–283.
[13] Mao T T, Jiang Z M, Han Y P, Teng W L, Zhao X, Li W B. Identification of quantitative trait loci underlying seed protein and oil contents of soybean across multi-genetic backgrounds and environments., 2013, 132: 630–641.
[14] Chung J, Babka H L, Graef G L, Staswicka P E, Lee D J, Cregan P B, Shoemaker R C, Specht J E. The seed protein, oil, and yield QTL on soybean linkage group I., 2003, 43: 1053–1067.
[15] 魏荷, 王金社, 卢为国. 大豆籽粒蛋白质含量分子遗传研究进展. 中国油料作物学报, 2015, 37: 394–400.
Wei H, Wang J S, Lu W G. Advances in molecular genetics of soybean grain protein content., 2015, 37: 394–400 (in Chinese with English abstract).
[16] 郭方亮. 大豆7S与11S球蛋白亚基缺失品系的鉴定与品质评价. 东北农业大学硕士学位论文, 黑龙江哈尔滨, 2019.
Guo F L. Identification and Quality Evaluation of Soybean 7S and 11S Globulin Subunit Deletion Strains. MS Thesis of Northeast Agricultural University, Harbin, Heilongjiang, China, 2019 (in Chinese with English abstract).
[17] 武阳春, 郭兵福, 谷勇哲, 栾晓燕, 邱红梅, 刘鑫磊, 李海燕, 邱丽娟. 大豆蛋白含量新位点的定位. 植物遗传资源学报, 2021, 22: 139–148.
Wu Y C, Guo B F, Gu Y Z, Luan X Y, Qiu H M, Liu X L, Li H Y, Qiu L J. Localization of a new protein content locusin soybean., 2021, 22: 139–148 (in Chinese with English abstract).
[18] Yang H Y, Wang W B, He Q Y, Xiang S H, Tian D, Zhao T J, Gai J. Identifying a wild allele conferring small seed size, high protein content and low oil content using chromosome segment substitution lines in soybean., 2019, 132: 2793–2807.
[19] Zhang T F, Wu T T, Wang L W, Jiang B J, Zhen C X, Yuan S, Hou W S, Wu C X, Han T F, Sun S. A combined linkage and GWAS analysis identifies QTLs linked to soybean seed protein and oil content., 2019, 20: 19.
[20] 郭茜茜. 大豆子粒蛋白质积累与碳代谢关系的研究. 东北农业大学硕士学位论文, 黑龙江哈尔滨, 2010.
Guo X X. Study on the Relationship Between Protein Accumulation and Carbon Metabolism in Soybean Seeds. MS Thesis of Northeast Agricultural University, Harbin, Heilongjiang, China, 2010 (in Chinese with English abstract).
[21] Zhong Y S, Lu X D, Deng Z W, Lu Z Q, Fu M H. A 1232 bp upstream sequence of glutamine synthetase 1b fromis a root-preferential promoter sequence., 2021, 21: 14.
[22] 陈欢. 大豆籽粒不同发育时期基因表达谱的研究. 吉林农业大学博士学位论文, 吉林长春, 2012.
Chen H. Study on Gene Expression Profile of Soybean Grain at Different Development Stages. PhD Dissertation of Jilin Agricultural University, Changchun, Jilin, China, 2012 (in Chinese with English abstract).
[23] Wei Z Y, Pan T, Zhao Y Y, Song B H, Qiu L J. Rab5a and its gefs are involved in post-golgi trafficking of storage proteins in developing soybean cotyledon., 2019, 71: 808–822.
[24] Wolf W J, Briggs D R. Purification and characterization of the 11S component of soybean proteins., 1959, 85: 186–199.
[25] Hara-Nishimura I, Nishimura M. Proglobulin processing enzyme in vacuoles isolated from developing pumpkin cotyledons., 1987, 85: 440–445.
[26] Kirsch T, Paris N, Butler J M, Beevers L, Rogers J C. Purification and initial characterization of a potential plant vacuolar targeting receptor., 1994, 91: 3403–3407.
[27] Okita T W, Rogers J C. Compartmentation of proteins in the endomembrane system of plant cells., 1996, 47: 327–350.
[28] Nishizawa K, Maruyama N, Satoh R, Fuchikami Y, Higasa T, Utsumi S. A C-terminal sequence of soybean β-conglycinin α’ subunit acts as a vacuolar sorting determinant in seed cells., 2003, 34: 647–659.
[29] Rillingos S. Insights to the evolution of nucleobase-ascorbate transporters (NAT/NCS2 family) from the Cys-scanning analysis of xanthine permease XanQ., 2012, 3: 250–272.
[30] Karena E, Frillingos S. The role of transmembrane segment TM3 in the xanthine permease XanQ of., 2011, 286: 39595–39605.
[31] Amillis S, Kosti V, Pantazopoulou A, Mikros E, Diallinas G. Mutational analysis and modeling reveal functionally critical residues in transmembrane segments 1 and 3 of the uapa transporter., 2011, 411: 567–580.
[32] Gournas C, Papageorgiou I, Diallinas G. The nucleobase- ascorbate transporter (NAT) family: genomics, evolution, structure- function relationships and physiological role., 2008, 4: 404–416.
[33] Eberhardt R Y, Chang Y Y, Bateman A G, Axelrod A L, Hwang W C, Aravind L. Filling out the structural map of the NTF2-like superfamily., 2013, 327: 11.
[34] Guillen K D, Lorrain C, Tsan P, Barthe P, Hecker A. Structural genomics applied to the rust fungusreveals two candidate effector proteins adopting cystine knot and NTF2-like protein folds., 2019, 9: 18084.
[35] Carazo-Salas R E, Gruss O J, Mattaj I W, Karsenti E. Ran-GTP coordinates regulation of microtubule nucleation and dynamics during mitotic-spindle assembly., 2001, 3: 228–234.
[36] Hetzer M, Bilbaocortés D, Walther T C, Gruss O J, Mattaj I W. GTP hydrolysis by ran is required for nuclear envelope assembly., 2000, 5: 1013–1024.
[37] Zhang Q, Wang B, Wei J, Wang X, Han Q, Kang Z., a contributor for wheat resistance to the stripe rust pathogen., 2018, 123: 260–267.
[38] Yang J, Yu D, Shen S. Expression analyses of miRNA Up-MIR- 843 and its target genes in., 2020, 39: 27–34.
[39] 王婉, 韩德志, 闫洪睿, 栾晓燕, 王俊, 邱丽娟. 大豆高蛋白种质中引1106蛋白质含量的QTL分析. 植物遗传资源学报, 2020, 21: 130–138.
Wang W, Han D Z, Yan H R, Luan X Y, Wang J, Qiu L J. QTL analysis of protein content in soybean high-protein germplasm for citation 1106., 2020, 21: 130–138 (in Chinese with English abstract).
[40] 闫海波, 王艳, 赵琳, 韩英鹏, 李文滨, 王桂玲. 大豆蛋白和油分含量的QTL分析. 大豆科学, 2016, 35: 228–233.
Yan H B, Wang Y, Zhao L, Han Y P, Li W B, Wang G L. QTL analysis associated with protein and oil content in soybean., 2016, 35: 228–233 (in Chinese with English abstract).
[41] Huang J H, Ma Q B, Cai Z D, Xia Q J, Li S X, Jia J, Chu L, Lian T X, Nian H, Cheng Y B. Identification and mapping of stable QTLs for seed oil and protein content in soybean [(L.) Merr.]., 2020, 68: 6448–6460.
[42] Kaleri A, Li L, Zhang Y, Liu W, Jiang C, Zhang Y, Liu C, Kaleri A H, Nizamani M M, Mehmood A, Bahadur S, Li W X, Ning H. Recognition of QTL for seed protein and oil content in two soybean recombinant inbred lines populations., 2021, 31: 1669–1685.
Fine mapping ofrelated to high protein content in soybean
YANG Shuo1,3,**, WU Yang-Chun4,**, LIU Xin-Lei2, TANG Xiao-Fei2, XUE Yong-Guo2, CAO Dan2, WANG Wan3, LIU Ting-Xuan3, QI Hang3, LUAN Xiao-Yan2,*, and QIU Li-Juan1,3,*
1College of Agriculture, Northeast Agricultural University, Harbin 150030, Heilongjiang, China;2Heilongjiang Academy of Agricultural Sciences, Harbin 150030, Heilongjiang, China;3College of Agriculture, Northeast Agricultural University, Harbin 150030, Heilongjiang, China;3National Key Facility for Gene Resources and Genetic Improvement / Key Laboratory of Crop Germplasm Utilization, Ministry of Agriculture and Rural Affairs / Institute of Crop Sciences, Chinese Academy of Agricultural Sciences, Beijing 100081, China;4College of Life Sciences, Jilin Agricultural University, Changchun 130000, Jinlin, China
Protein content is a crucial quality trait of soybean, which is controlled by multiple genes. It is of great significance to locate soybean protein content-related loci and mine candidate genes for directional breeding of soybean varieties with high protein content. In this study, an F2population consisting of 265 individual plants was constructed by crossing the excellent variety Heinong 88 as the female parent with the high-protein germplasm P73-6B as the male parent. The genotypes of F2population were identified by using high-density SNP chip of “ZDX1” and the physical map was constructed. Combined with the protein content phenotypic data, the initial mapping interval of a 2.46 Mb QTL was located on chromosome 20 using the IciMapping 4.2 software. Using 11 polymorphic SSR markers screened out, the mapping range was narrowed from 2.46 Mb to 100.8 kb. Adding four SNP markers (Gm20_28349696, Gm20_30805913, Gm20_31341532, and Gm20_31483719), the interval was further reduced to 95.8 kb. The relative expression levels of the four genes contained in the interval in nine different tissues in both databases Phytozome v13.1 and PPRD RNA-seq yielded two candidate genes (and). These results provide a theoretical basis for soybean protein content gene cloning and protein regulation mechanism research, as well as elite material and molecular marker for breeding high protein soybean.
soybean; protein content; QTL mapping; candidate genes
10.3724/SP.J.1006.2023.24015
本研究由国家自然科学基金项目(31960408), 中央级公益性科研院所基本科研业务费专项(S2022ZD02)和中国农业科学院科技创新工程项目资助。
This study was supported by the National Natural Science Foundation of China (31960408), the Central Public-interest Scientific Institution Basal Research Fund (S2022ZD02), and the Agricultural Science and Technology Innovation Program.
邱丽娟, E-mail: qiulijuan@caas.cn
**同等贡献(Contributed equally to this work)
杨硕, E-mail: 857813782@qq.com
2022-01-10;
2022-06-07;
2022-07-08.
URL: https://kns.cnki.net/kcms/detail/11.1809.S.20220707.1133.005.html
This is an open access article under the CC BY-NC-ND license (http://creativecommons.org/licenses/by-nc-nd/4.0/)
猜你喜欢 表型区间位点 你学会“区间测速”了吗中学生数理化·八年级物理人教版(2022年9期)2022-10-24镍基单晶高温合金多组元置换的第一性原理研究上海金属(2021年6期)2021-12-02CLOCK基因rs4580704多态性位点与2型糖尿病和睡眠质量的相关性昆明医科大学学报(2021年3期)2021-07-22基于网络公开测序数据的K326烟草线粒体基因组RNA编辑位点的鉴定与分析烟草科技(2021年6期)2021-06-24全球经济将继续处于低速增长区间中国外汇(2019年13期)2019-10-10一种改进的多聚腺苷酸化位点提取方法电脑知识与技术(2018年19期)2018-11-01建兰、寒兰花表型分析现代园艺(2017年21期)2018-01-03miR-363-3p表达异常对人前列腺癌细胞生物学表型的影响中国男科学杂志(2016年5期)2016-12-01区间对象族的可镇定性分析北京信息科技大学学报(自然科学版)(2016年6期)2016-02-27GABABR2基因遗传变异与肥胖及代谢相关表型的关系中国康复理论与实践(2015年10期)2015-12-24猜你喜欢
- 2024-01-20 有关于第五次全国经济普查统计重点业务综合培训大会上讲话(完整文档)
- 2024-01-20 “严纪律、转作风、保安全、树形象”专题学习教育活动通知(完整文档)
- 2024-01-20 2024XX区住房城乡建设工作情况汇报
- 2024-01-20 2024高校思政教育交流材料:善用反腐败斗争这堂“大思政课”(精选文档)
- 2024-01-20 2024年主题教育专题党课辅导报告,(4)
- 2024-01-20 关于赴某地学习考察地方立法工作情况报告(范文推荐)
- 2024-01-20 2024年度关于增强党建带团建工作实效对策与建议(精选文档)
- 2024-01-20 教师演讲稿:春风化雨育桃李,,潜心耕耘满芬芳(全文)
- 2024-01-20 主题教育第二阶段来了
- 2024-01-20 2024年度关于到信访局实践锻炼个人总结【完整版】
- 搜索
-
- 打赌输了任人处理作文1000字7篇 05-12
- 当代大学生在全面建设社会主义现代化强 05-12
- 全面建成社会主义现代化强国的战略安排 03-10
- 个人廉洁自律方面存在的问题及整改措施 05-12
- 谈谈青年大学生在中国式现代化征程上的 05-12
- 2022年党支部第一议题会议记录(全文完 11-02
- 作为青年大学生如何肩负时代责任6篇 05-12
- 村党组织建设现状及工作亮点存在问题与 05-12
- 全面从严治党,自我革命重要论述研讨会 05-12
- 产业工人队伍建设改革(全文完整) 10-31
- 11-25国庆70周年庆典晚会 庆典晚会串词
- 11-25办公室礼仪的十大原则 浅谈办公室的电话礼仪
- 01-17用心灵轻轻地歌唱_心灵的歌唱
- 01-17也许你不是我一生的唯一|也许不是我
- 01-17爱了,请珍惜;不爱,趁早放手|爱就珍惜不爱就放手
- 01-17岁月带走的是记忆,但回忆会越来越清晰|有趣又有深意的句子
- 01-17曾经的美好只是曾经,我只想珍惜身边的人|我只想珍惜你
- 01-18从容不惊 [学会笑眼去看世界,不惊不乍,淡定从容]
- 02-03当代大学生学习态度调查报告
- 02-03常用护患英语会话
- 标签列表
